Este articulo lo escribimos en el año 2001 cuando terminamos nuestro primer semestre del curso de Ciencias Genómicas en el CIFN (ahora CCG). Se publicó en una revista que teníamos por aquellos tiempos en la facultad y lo publico aqui ahora pues a pesar del paso del tiempo no ha perdido validez y tiene valor divulgatorioLA GENÓMICA, DESCUBRIENDO EL UNIVERSO GENÉTICO
Ana Laura Ramos Vega, Edgardo Sepúlveda S.H., David Romero Camarena
El genoma es el contenido total de ADN en una célula, que en el caso de los eucariontes está representado por el genoma nuclear y el mitocondrial; en las plantas también el de los cloroplastos. Tan solo en los humanos el genoma nuclear está formado por 3 000 000 000 de pares de bases divididas en 24 moléculas lineares de ADN llamadas cromosomas; de éstos, 22 son autosomas y 2 son cromosomas sexuales. Solamente una pequeña fracción de éstos 3 mil millones de bases, estimada en tan solo el 3% (90 000 000 bases), contribuye con la información para los 80 000 genes que se cree que posee nuestra especie.
Cuando se demostró, allá por 1944 que el ADN era la molécula que contenía toda la información para llevar a cabo las funciones biológicas de un ser vivo -e incluso para el desarrollo de otro igual- fue muy obvio que al comprender y poder leer el lenguaje en que se guardaba esa información podríamos entender la vida de una manera más profunda. Durante treinta años los científicos trabajaban a ciegas y cuando por fin pudieron leerla lo hacían de una forma limitada, lenta y laboriosa. Sin embargo hoy, gracias al ingenio y trabajo del hombre, poseemos una herramienta tan poderosa que nos ha permitido acceder en tan solo cinco años a una información enorme y tan compleja que resulta dificil imaginar sus alcances. Esta herramienta es la genómica, que ayudada por procedimientos químicos, biológicos, matemáticos y computacionales abre las puertas a la secuenciación y caracterización de genomas completos, cuyo análisis revolucionará sin lugar a dudas todos los aspectos del conocimiento humano.
La SecuenciaciónA finales de los 70’s fueron publicados dos métodos rápidos y eficientes de secuenciación de ADN: el de degradación química de Maxam y Gilbert y el de terminación de cadena ideado por Sanger. Este último es la base de los metodos de secuenciación usados actualmente y por lo tanto es del que nos ocuparemos. El método se basa en que se pueden separar cadenas simples de ADN (hélices sencillas no dobles) que difieran de tamaño en tan solo una base al analizarlas en una electroforesis. Estas moléculas se obtienen en una reacción in vitro de síntesis de ADN, en donde una enzima, la polimerasa de ADN sintetiza la cadena complementaria a la molécula a secuenciar a partir de 4 deoxiribonucleótidos trifosfatados (dATP, dGTP, dCTP dTTP) los cuales servirán como substratos. Adicionalmente se provee una cantidad limitante de uno de estos nucleótidos modificado en su carbono 3 substituyendo el grupo OH por un H (dideoxiribonucleótidos), los cuales al ser incorporados a la cadena complementaria bloquean la elongación de ésta, deteniendo el proceso. Así, se obtienen moléculas de diferentes tamaños que nos marcan el lugar donde se encuentra la base. Esta reacción se lleva a cabo 4 veces con los diferentes nucleótidos de manera que obtenemos la posición de cada uno de ellos en la cadena.
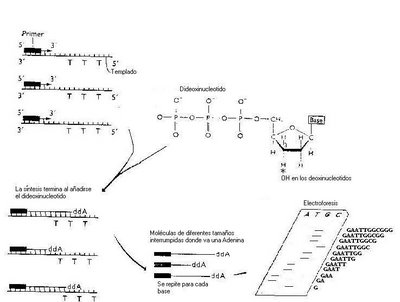
(Click en la imagen para agrandar en ventana nueva)
Actualmente el proceso está automatizado y se pueden secuenciar moléculas de hasta 1000 bases en longitud en unas cuantas horas. Como un genoma pequeño se mide por lo menos en términos de centenares de Kilobases, este debe ser partido en trozos, ya sea con enzimas o por métodos físicos y secuenciados por partes que posteriormente son ensamblados por traslape de sus secuencias. Como hay que asegurarse de que en los trozos esté representado todo el genoma se hacen los cortes por lo menos 3 veces en sitios distintos y se secuencian. Es así que para secuenciar el genoma humano (3 000 000 000 de pares de bases) se debieron secuenciar aproximadamente 9 000 000 000 de bases por cada cadena del ADN para un total de 18 000 000 000 de bases que representan 45 millones de secuenciaciones.
La secuencia en sí no descifra toda la información contenida en el genoma, solamente es el primer paso. Posteriormente se deben integrar todos los fragmentos ya secuenciados para conocer su estructura física; este gran rompecabezas no solo está formado por piezas únicas, a lo largo de todo genoma se presentan repeticiones de secuencias de diversos tipos (satélites, microsatélites, minisatélites). La función de estas secuencias repetidas hasta ahora es muy poco conocida o totalmente un misterio. Si bien su posible función constituye una interrogante científica muy relevante, su mera existencia plantea un problema técnico formidable. Al momento de querer integrar una secuencia traslapante no siempre es seguro que ésta se ubique en el lugar exacto por lo que el armado se realiza solo después de comparar la secuencia con otras que se encuentren cercanas al lugar hipotético y probarlo con todas las posiciones posibles, este proceso se lleva a cabo asistido por computadora dado el tamaño de la información a procesar, sin embargo la intervención humana es indispensable pues no hay máquina capaz de igualar el raciocinio humano.
Despues de la SecuenciaciónLa verdadera importancia de obtener la secuencia de un genoma radica en tener la posibilidad de descubrir y entender la función de los genes. Tradicionalmente se identificaba una proteína por su función para después dilucidar de que gen provenía; actualmente a partir del genoma secuenciado, esto se puede hacer al revés. Obviamente para esto tenemos que identificar un gen, esto puede ser de tres maneras que nos abren nuevas posibilidades:
1.- Si queremos encontrar en un organismo un gen conocido ya en otro, bastará con comparar su secuencia con el genoma para identificar la más similar a éste, si ésta reúne las características de un gen puede enfocarse el estudio en él para saber si cumplen con la misma función. Esto tiene otras implicaciones positivas, al encontrar homología entre dos genes podemos estudiar si los dos organismos provienen de un ancestro común y entender parte de su historia evolutiva, es decir si los organismos divergieron antes de el gen o el gen antes de los organismos.
2.- Si conocemos una proteína, a partir de su secuencia de aminoácidos podemos inferir la de nucleótidos y por el método anterior localizar el gen en el genoma. Esto presenta cierta dificultad, ya que la secuencia de aminoácidos no siempre es una representación fiel del gen, pues el ARNm sintetizado a partir de éste tiene secciones llamadas intrones que son retiradas antes de salir del núcleo para su traducción en eucariontes además de que la proteína tambien sufre de la edición de algunos aminoácidos después de su traducción. También pueden existir copias incompletas no funcionales de los genes, llamadas pseudogenes que tienen amplia homología con el gen funcional por lo que pueden distraer los resultados. Las aplicaciones de esto son prometedoras, a partir de la proteína causante de una enfermedad podemos identificar el gen del que se traduce e investigar como inhibirlo o hacer lo mismo con el de una proteína esencial para un agente patógeno.
3.- La tercera posibilidad es la más aventurera porque parte de cero. En un gen está estructurado lo que se define como un marco de lectura abierto (orf por sus siglas en inglés) que es una serie de codones que especifican para aminoácidos que formarán parte de la proteína, además de que incluyen los sitios a partir de los cuales iniciará y terminará la traducción. Fuera de este marco se encuentran los sitios de reconocimiento para las enzimas involucradas en la transcripción del gen y por lo tanto en la regulación de éste. Se pueden localizar en los genomas los orfs e incluso inferir la actividad de la proteína para la que podrían codificar al analizar su secuencia por los dominios de aminoácidos, por ejemplo si la secuencia sugiere un dominio de estructura terciaria hélice-vuelta-hélice podemos inferir que esta proteína interactúa con el ADN; de esta forma se puede enfocar la atención a la zonas de interés con un gran ahorro de tiempo.
El RetoLo cierto es que aquí no termina todo, las aplicaciones prácticas son útiles y reconocemos el avance que esto significará para áreas como la médica sin embargo el conocimiento genómico involucra retos adicionales de gran importancia. A partir de la inmensa cantidad de genes identificados y predichos en los ya 35 genomas secuenciados (
este dato ha sido actualizado aquí) y los genes conocidos previamente en otros organismos, se construyen bases de datos. Con éstas se pueden hacer análisis por comparación e identificación de patrones y peculiaridades en las secuencias para genes con función similar y entre genomas, lo que con ayuda de análisis matemáticos permitirá establecer métodos de predicción de genes, operones y funciones tanto de las proteínas traducidas como de la regulación de su traducción y la estructura del genoma. Recordemos que solo una pequeña parte de los genomas es codificante, pero eso no significa que el resto no sirva para nada o tenga que servir para algo, existen pseudogenes, intrones y secuencias repetidas de unas cuantas bases de largo o de cientos de ellas y elementos como los transposones que son secuencias capaces de hacerse saltar a ellas mismas o a una copia a diferentes puntos del genoma y que pueden o no ser codificantes, genes dentro de los intrones y genes que pueden traducirse en diferentes proteínas según sea necesario y muchas otras cosas que ni siquiera podemos comprender ya que tenemos más información de la que podemos manejar. En tiempos venideros, corresponderá a equipos multidisciplinarios, compuestos de biólogos, físicos, matemáticos e informáticos de nuestra generación el desentrañar los secretos que involucra la molécula responsable de la herencia biológica, el ADN y es que la comprensión del aspecto funcional, estructural y evolutivo del genoma así como su aplicación entrañará, a no dudarse, un cambio en la concepción que tiene el hombre de la naturaleza, de la vida y de él mismo, lo que nos llevará a una nueva etapa en la historia de la humanidad.
INTERNET-
Centro de Ciencias Genómicas, UNAM-
GenBank-
Institute for Genome Research -Protein Domain Database GLOSARIOAutosoma.- Cromosoma que no es sexual.
Cloroplasto.- Organelo membranoso en células autótrofas donde se lleva a cabo la
fotosíntesis.
Gen .- Segmento del ADN que codifica para una proteína.
Mitocondria.- Organelo membranoso en células eucariontes donde se lleva a cabo la Oxidación.
Núcleo.- Organelo membranoso en célulalas eucariontes que contiene el ADN
Traducción.- Proceso mediante el cual los tripletes de bases del ADN son traducidos a aminoácidos para formar una proteína.
Transcripción.- Síntesis de una molécula de ARN a partir de una de ADN
